Структурная вариация - Structural variation
Геномные структурные вариации это изменение структуры организма хромосома. Он состоит из множества разновидностей вариаций геном одного разновидность, и обычно включает микроскопический и субмикроскопический типы, такие как удаления, дублирования, варианты номера копии, прошивки, инверсии и транслокации. Первоначально изменение структуры влияет на длину последовательности от 1 кбайт до 3 Мбайт, которая больше, чем SNP и меньше чем хромосомная аномалия (хотя определения частично совпадают).[1] Тем не менее, операционный диапазон структурных вариантов расширился и теперь включает события> 50 б.п.[2]. Определение структурной изменчивости ничего не говорит о частоте или фенотипических эффектах. Многие структурные варианты связаны с генетические заболевания, однако многие из них - нет.[3][4] Недавнее исследование SV показывает, что SV труднее обнаружить, чем SNP. Примерно 13% генома человека определено как структурно вариантное в нормальной популяции, и существует не менее 240 генов, которые существуют как гомозиготный удаление полиморфизмы в человеческих популяциях, предполагая, что эти гены не обязательны для людей.[4] Быстро накапливающиеся данные показывают, что структурные вариации могут включать миллионы неоднородных нуклеотидов в каждом геноме и, вероятно, вносят важный вклад в разнообразие людей и восприимчивость к заболеваниям.
Микроскопические структурные вариации
Микроскопия означает, что его можно обнаружить с помощью оптические микроскопы, такие как анеуплоидии, маркерная хромосома, грубые перестройки и вариации в размере хромосом.[5][6] Считается, что частота в человеческой популяции недооценена из-за того, что некоторые из них на самом деле нелегко идентифицировать. По предполагаемым данным, эти структурные аномалии существуют у 1 из 375 живорождений.[7]
Субмикроскопические структурные вариации
Субмикроскопические варианты структуры гораздо труднее обнаружить из-за их небольшого размера. Первое исследование 2004 г., в котором использовались ДНК-микрочипы смог обнаружить десятки генетических места что выставлено изменение количества копий, удаления и дублирование, больше 100 килобазы в геноме человека.[8] Однако к 2015 году исследования по секвенированию всего генома могут выявить около 5000 структурных вариантов размером всего 100 пар оснований это примерно 20 мегабазы в каждом отдельном геноме.[3][4] Эти структурные варианты включают делеции, тандемные дупликации, инверсии, вставки мобильных элементов. Частота мутаций также намного выше, чем у микроскопических структурных вариантов, по оценкам двух исследований в 16% и 20% соответственно, оба из которых, вероятно, занижены из-за проблем с точным обнаружением структурных вариантов.[3][9] Также было показано, что генерация спонтанных структурных вариантов значительно увеличивает вероятность генерации дальнейших спонтанных однонуклеотидные варианты или инделы в пределах 100 килобазы события структурной вариации.[3]
Вариант номера копии
Вариация числа копий (CNV) - это большая категория структурных вариаций, которая включает вставки, удаления и дублирование. В недавних исследованиях вариации числа копий проверяются на людях, не страдающих генетическими заболеваниями, с использованием методов, которые используются для количественного генотипирования SNP. Результаты показывают, что 28% подозрительных областей у людей действительно содержат вариации количества копий.[10][11] Кроме того, CNV в геноме человека влияют на большее количество нуклеотидов, чем Однонуклеотидный полиморфизм (SNP). Также стоит отметить, что многие CNV не находятся в кодирующих областях. Поскольку CNV обычно вызваны неравная рекомбинация, широко распространенные подобные последовательности, такие как ЛИНИИ и СИНУСЫ может быть обычным механизмом создания CNV.[12][13]
Инверсия
Известно несколько инверсий, связанных с болезнями человека. Например, повторяющаяся инверсия 400kb в гене фактора VIII является частой причиной гемофилия А,[14] и меньшие инверсии, влияющие на идунорат 2-сульфатаза (IDS) вызовет Синдром Хантера.[15] Дополнительные примеры включают Синдром ангельмана и Синдром Сотоса. Однако недавние исследования показывают, что у одного человека может быть 56 предполагаемых инверсий, поэтому инверсии, не связанные с болезнью, встречаются чаще, чем предполагалось ранее. Также в этом исследовании указано, что точки останова инверсии обычно связаны с сегментарными дупликациями.[16] Одна инверсия 900 кб в хромосома 17 находится под положительный выбор и прогнозируется увеличение его частоты среди европейского населения.[17]
Другие варианты конструкции
Могут возникнуть более сложные структурные варианты, включающие комбинацию вышеперечисленного в одном событии.[3] Наиболее распространенным типом сложных структурных вариаций являются нетандемные дупликации, когда последовательность дублируется и вставляется в инвертированной или прямой ориентации в другую часть генома.[3] Другие классы сложных структурных вариантов включают делеции-инверсии-делеции, дупликации-инверсии-дупликации и тандемные дупликации с вложенными делециями.[3]Это также загадочные транслокации и сегментарная однопородная дисомия (UPD). Сообщений об этих вариациях становится все больше, но их труднее обнаружить, чем традиционные вариации, потому что эти варианты сбалансированы и основаны на массивах или ПЦР методы не могут их найти.[18]
Структурная изменчивость и фенотипы
Немного генетические заболевания подозреваются, что они вызваны структурными изменениями, но связь не очень определена. Невозможно разделить эти варианты на два класса: «нормальный» или «болезненный», потому что фактический результат одного и того же варианта также будет отличаться. Кроме того, некоторые из вариантов действительно положительно отобраны для (упомянутого выше). Серия исследований показала, что нарушение генов спонтанный (de novo) CNVs разрушают гены примерно в четыре раза чаще при аутизме, чем в контрольной группе, и составляют примерно 5–10% случаев.[3][19][20][21][22] Унаследованные варианты также вызывают около 5–10% случаев аутизма.[3]
Структурные вариации также играют свою роль в популяционной генетике. Различная частота одной и той же вариации может использоваться в качестве генетической метки для вывода о взаимосвязи между популяциями в разных областях. Полное сравнение структурных вариаций человека и шимпанзе также показало, что некоторые из них могут быть зафиксированы у одного вида из-за его адаптивной функции.[23] Есть также делеции, связанные с сопротивлением против малярия и СПИД.[24][25] Также считается, что некоторые сильно изменчивые сегменты вызваны балансирующим отбором, но есть также исследования, опровергающие эту гипотезу.[26]
База данных структурных вариаций
Некоторые браузеры генома и биоинформатический базы данных содержат список структурных вариаций генома человека с акцентом на CNV и могут отображать их на странице просмотра генома, например, Браузер генома UCSC.[27] Под страницей просмотра части генома можно включить "Common Cell CNVs" и "Structural Var". В NCBI есть специальная страница [28] для структурных вариаций. В этой системе показаны как «внутренние», так и «внешние» координаты; они оба не являются фактическими точками останова, но предполагают минимальный и максимальный диапазон последовательности, на которую влияет структурная вариация. Типы классифицируются как вставка, потеря, усиление, инверсия, LOH, вывернутый, transchr и UPD.[нужна цитата ]
Методы обнаружения
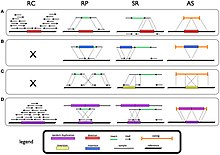
Были разработаны новые методы анализа генетической структурной изменчивости человека с высоким разрешением. Методы, используемые для тестирования генома, являются либо целенаправленными, либо общегеномными. Для полногеномных тестов подходы сравнительной гибридизации генома на основе массивов обеспечивают наилучшее сканирование всего генома для поиска новых вариантов числа копий.[30] В этих методах используются фрагменты ДНК, которые помечены из интересующего генома и гибридизуются с другим геномом, помеченным по-другому, в массивы с пятнами клонированных фрагментов ДНК. Это показывает различия в количестве копий между двумя геномами.[30]
Для целевых исследований генома лучшие анализы для проверки конкретных участков генома в первую очередь основаны на ПЦР. Лучше всего из методов, основанных на ПЦР, является количественная полимеразная цепная реакция в реальном времени (КПЦР).[30] Другой подход состоит в том, чтобы специально проверить определенные области, которые окружают известные сегментные дублирования, поскольку они обычно являются областями изменения количества копий.[30] Метод генотипирования SNP, который предлагает независимые интенсивности флуоресценции для двух аллелей, может использоваться для нацеливания на нуклеотиды между двумя копиями сегментарной дупликации.[30] Отсюда можно наблюдать увеличение интенсивности одного из аллелей по сравнению с другим.
С развитием секвенирование следующего поколения (NGS) было сообщено о четырех классах стратегий для обнаружения структурных вариантов с данными NGS, каждый из которых основан на шаблонах, которые являются диагностическими для разных классов SV.[29][31][32]
- Методы глубины чтения или подсчета чтения предполагают случайное распределение (например, распределение Пуассона ) из читает от секвенирования. Отклонение от этого распределения исследуется для обнаружения дубликатов и удалений. В областях с дублированием будет отображаться более высокая глубина чтения, а в областях с удалением - меньшая глубина чтения.
- Сплит-методы чтения позволяют обнаруживать вставки (в том числе мобильный элемент вставки) и делеции вплоть до разрешения одной пары оснований. Наличие SV идентифицируют по прерывистому выравниванию с эталонным геномом. Пробел в прочитанном означает удаление, а в ссылках - вставку.
- Методы чтения пар проверяют длину и ориентацию чтения парного конца. Пары считывания дальше друг от друга, чем ожидалось, указывают на удаление, в то время как пары, найденные слишком близко, указывают на вставку.
- De novo Сборка последовательности может применяться с достаточно точными считываниями. Однако использование этого метода на практике ограничено из-за недостаточной длины чтения последовательности.
Смотрите также
Рекомендации
- ^ Феук, Ларс; Карсон, Эндрю Р .; Шерер, Стивен В. (2006). «Структурные вариации в геноме человека». Природа Обзоры Генетика. 7 (2): 85–97. Дои:10.1038 / nrg1767. PMID 16418744. S2CID 17255998.
- ^ Алкан, банка; Коу, Брэдли П.; Эйхлер, Эван Э. (01.03.2011). «Открытие структурных вариаций генома и генотипирование». Природа Обзоры Генетика. 12 (5): 363–376. Дои:10.1038 / nrg2958. ISSN 1471-0056. ЧВК 4108431. PMID 21358748.
- ^ а б c d е ж грамм час я Брандлер, Уильям М .; Антаки, Дэнни; Гуджрал, Мадхусудан; Нур, Амина; Розанио, Габриэль; Чапмен, Тимоти Р .; Barrera, Daniel J .; Линь Гуань Нин; Малхотра, Дирадж; Уоттс, Аманда С.; Вонг, Лоуренс С .; Эстабилло, Джаспер А .; Гадомский, Тереза Э .; Хонг, Оань; Фахардо, Карин В. Фуэнтес; Бхандари, Абхишек; Оуэн, Рениус; Бон, Майкл; Юань, Джеффри; Соломон, Терри; Мойзис, Александра Г .; Maile, Michelle S .; Сандерс, Стефан Дж .; Райнер, Гейл Э .; Vaux, Keith K .; Стром, Чарльз М .; Чжан, Канг; Muotri, Alysson R .; Акшумофф, Наташа; Leal, Suzanne M .; Пирс, Карен; Куршн, Эрик; Якучева, Лилия М .; Корселло, Кристина; Себат, Джонатан (24 марта 2016 г.). «Частота и сложность структурной мутации De Novo при аутизме». Американский журнал генетики человека. 98 (4): 667–79. Дои:10.1016 / j.ajhg.2016.02.018. ЧВК 4833290. PMID 27018473.
- ^ а б c Судмант, Питер Х .; Рауш, Тобиас; Гарднер, Юджин Дж .; Хандакер, Роберт Э .; Абызов Алексей; Хаддлстон, Джон; Чжан, Ян; Йе, Кай; Джун, Гу; Си-Ян Фриц, Маркус; Konkel, Miriam K .; Мальхотра, Анкит; Stütz, Adrian M .; Ши, Синхуа; Паоло Казале, Франческо; Чен, Цзимин; Хормоздиари, Ферейдун; Даяма, Гарги; Чен, Кен; Малиг, Майка; Chaisson, Mark J. P .; Вальтер, Клаудиа; Мейерс, Саша; Кашин, Сева; Гаррисон, Эрик; Аутон, Адам; Lam, Hugo Y. K .; Жасмин Му, Синьмэн; Алкан, банка; Антаки, Дэнни; Пэ, Тэджон; Сервейра, Элиза; Чайнс, Питер; Чонг, Зечен; Кларк, Лаура; Даль, Элиф; Дин, Ли; Эмери, Сара; Фань, Сиань; Гуджрал, Мадхусудан; Кахведжи, Фатьма; Кидд, Джеффри М .; Kong, Yu; Ламейер, Эрик-Вуббо; Маккарти, Шейн; Фличек, Пол; Гиббс, Ричард А .; Март, Габор; Мейсон, Кристофер Э .; Менелау, Андроники; Музны, Донна М .; Нельсон, Брэдли Дж .; Нур, Амина; Пэрриш, Николас Ф .; Пендлтон, Мэтью; Куитадамо, Эндрю; Редер, Бенджамин; Schadt, Eric E .; Романович, Мэллори; Шлаттль, Андреас; Себра, Роберт; Шабалин, Андрей А .; Унтергассер, Андреас; Уокер, Джелин А .; Ван, Мин; Ю, Фули; Чжан, Чэншэн; Чжан, Цзин; Чжэн-Брэдли, Сянцюнь; Чжоу, Wanding; Зихнер, Томас; Себат, Джонатан; Batzer, Mark A .; МакКэрролл, Стивен А .; Миллс, Райан Э .; Герштейн, Марк Б.; Башир Али; Стегл, Оливер; Дивайн, Скотт Э .; Ли, Чарльз; Эйхлер, Эван Э.; Корбель, Ян О. (30 сентября 2015 г.). «Интегрированная карта структурных вариаций в 2 504 геномах человека». Природа. 526 (7571): 75–81. Bibcode:2015Натура 526 ... 75.. Дои:10.1038 / природа15394. ЧВК 4617611. PMID 26432246.
- ^ Райх, Дэвид Э .; Шаффнер, Стивен Ф .; Дейли, Марк Дж .; Маквин, Гил; Малликин, Джеймс С .; Хиггинс, Джон М .; Рихтер, Дэниел Дж .; Лендер, Эрик С.; Альтшулер, Давид (2002). «Вариации последовательности генома человека и влияние истории генов, мутации и рекомбинации». Природа Генетика. 32 (1): 135–42. Дои:10,1038 / ng947. PMID 12161752. S2CID 16822751.
- ^ Грипенберг, Улла (1964). «Изменение размера и ориентация хромосомы Y человека». Хромосома. 15 (5): 618–29. Дои:10.1007 / BF00319995. PMID 14333154. S2CID 26549548.
- ^ Wyandt, H.E .; Тонк, В. С. (2004). Атлас гетероморфизмов хромосом человека. Нидерланды: Kluwer Academic. ISBN 978-90-481-6296-3.[страница нужна ]
- ^ Себат, Дж. (23 июля 2004 г.). «Полиморфизм большого числа копий в геноме человека». Наука. 305 (5683): 525–528. Bibcode:2004Наука ... 305..525С. Дои:10.1126 / science.1098918. PMID 15273396.
- ^ Kloosterman, Wigard P .; Francioli, Laurent C .; Хормоздиари, Ферейдун; Маршалл, Тобиас; Hehir-Kwa, Jayne Y .; Абделлауи, Абдель; Ламейер, Эрик-Вуббо; Moed, Matthijs H .; Коваль, Вячеслав; Ренкенс, Иво; van Roosmalen, Markus J .; Арп, Паскаль; Karssen, Lennart C .; Коу, Брэдли П.; Хандакер, Роберт Э .; Сучиман, Эка Д .; Куппен, Эдвин; Тхунг, Джие Тджван; Маквей, Митч; Вендл, Майкл С.; Уиттерлинден, Андре; van Duijn, Cornelia M .; Swertz, Morris A .; Вейменга, Сиска; van Ommen, GertJan B .; Slagboom, P. Eline; Boomsma, Dorret I .; Шёнхут, Александр; Эйхлер, Эван Э.; de Bakker, Paul I.W .; Йе, Кай; Гурьев, Виктор (июнь 2015). «Характеристики структурных изменений de novo в геноме человека». Геномные исследования. 25 (6): 792–801. Дои:10.1101 / гр.185041.114. ЧВК 4448676. PMID 25883321.
- ^ Sebat, J .; Лакшми, Б; Troge, J; Александр, J; Янг, Дж; Лундин, П; Månér, S; Масса, Н; и другие. (2004). «Полиморфизм большого числа копий в геноме человека». Наука. 305 (5683): 525–8. Bibcode:2004Наука ... 305..525С. Дои:10.1126 / science.1098918. PMID 15273396.
- ^ Иафрате, Джон; Феук, Ларс; Ривера, Мигель Н; Листевник, Марк Л; Донахью, Патрисия К.; Ци, Инь; Шерер, Стивен В.; Ли, Чарльз (2004). «Обнаружение крупномасштабных вариаций в геноме человека». Природа Генетика. 36 (9): 949–51. Дои:10,1038 / ng1416. PMID 15286789.
- ^ Лупски, Джеймс Р. (2010). «Ретротранспозиция и структурные изменения в геноме человека». Клетка. 141 (7): 1110–2. Дои:10.1016 / j.cell.2010.06.014. PMID 20602993. S2CID 2047696.
- ^ Лам, Хьюго Ю.К .; Му, Жасмин Синьмэн; Штутц, Адриан М; Танзер, Андреа; Кейтинг, Филип Д.; Снайдер, Майкл; Ким, Филип М; Корбель Ян О; Герштейн, Марк Б. (2010). «Анализ структурных вариантов с разрешением нуклеотидов с использованием BreakSeq и библиотеки точек останова». Природа Биотехнологии. 28 (1): 47–55. Дои:10.1038 / nbt.1600. ЧВК 2951730. PMID 20037582.
- ^ Лакич, Делия; Kazazian, Haig H .; Антонаракис, Стилианос Э .; Гитшиер, Джейн (1993). «Инверсии, разрушающие ген фактора VIII, являются частой причиной тяжелой гемофилии А». Природа Генетика. 5 (3): 236–41. Дои:10.1038 / ng1193-236. PMID 8275087. S2CID 25636383.
- ^ Бондесон, Мэр-Луиза; Даль, Никлас; Мальмгрен, Елена; Kleijer, Wim J .; Tönnesen, Tönne; Карлберг, Бритт-Мари; Петтерссон, Ульф (1995). «Инверсия гена IDS в результате рекомбинации с последовательностями, связанными с IDS, является общей причиной синдрома Хантера». Молекулярная генетика человека. 4 (4): 615–21. Дои:10,1093 / чмг / 4.4.615. PMID 7633410.
- ^ Тузун, Эрай; Шарп, Эндрю Дж; Бейли, Джеффри А; Каул, Раджиндер; Моррисон, В. Энн; Pertz, Lisa M; Хауген, Эрик; Хайден, Хиллари; и другие. (2005). «Мелкомасштабные структурные вариации генома человека». Природа Генетика. 37 (7): 727–32. Дои:10,1038 / ng1562. PMID 15895083. S2CID 14162962.
- ^ Стефанссон, Хрейнн; Хельгасон, Агнар; Торлейфссон, Гудмар; Штайнторсдоттир, Валгердур; Массон, Гисли; Барнард, Джон; Бейкер, Адам; Йонасдоттир, Аслауг; и другие. (2005). «Обычная инверсия при отборе у европейцев». Природа Генетика. 37 (2): 129–37. Дои:10,1038 / ng1508. PMID 15654335. S2CID 120515.
- ^ Сун, Винг-Кин ,. Алгоритмы секвенирования нового поколения. Бока-Ратон. п. 215. ISBN 978-1-4665-6551-7. OCLC 987790994.CS1 maint: лишняя пунктуация (ссылка на сайт) CS1 maint: несколько имен: список авторов (ссылка на сайт)
- ^ Sebat, J .; Лакшми, Б .; Malhotra, D .; Troge, J .; Lese-Martin, C .; Walsh, T .; Yamrom, B .; Yoon, S .; Красниц, А .; Kendall, J .; Leotta, A .; Оплаченный.; Zhang, R .; Lee, Y.-H .; Hicks, J .; Spence, S.J .; Ли, А. Т .; Пуура, К .; Lehtimaki, T .; Ledbetter, D .; Gregersen, P.K .; Bregman, J .; Sutcliffe, J. S .; Jobanputra, V .; Chung, W .; Warburton, D .; King, M.-C .; Скусе, Д .; Geschwind, D. H .; Gilliam, T. C .; Е, К .; Виглер, М. (20 апреля 2007 г.). «Сильная ассоциация мутаций числа копий De Novo с аутизмом». Наука. 316 (5823): 445–449. Bibcode:2007Sci ... 316..445S. Дои:10.1126 / science.1138659. ЧВК 2993504. PMID 17363630.
- ^ Пинто, Далила; Делаби, Эльза; Мерико, Даниэле; Барбоса, Мафалда; Мерикангас, Элисон; Клей, Ламбертус; Тирувахиндрапурам, Бхома; Сюй, Сяо; Зиман, Роберт; Ван, Чжуочжи; Vorstman, Jacob A.S .; Томпсон, Энн; Регана, Регина; Пилорж, Марион; Пеллеккья, Джованна; Pagnamenta, Alistair T .; Оливейра, Барбара; Marshall, Christian R .; Magalhaes, Tiago R .; Лоу, Дженнифер К .; Хау, Дженнифер Л .; Грисволд, Энтони Дж .; Гилберт, Джон; Дукетис, Эфтихия; Dombroski, Beth A .; Де Йонге, Марета В .; Куккаро, Майкл; Кроуфорд, Эмили Л .; Correia, Catarina T .; и другие. (Май 2014 г.). «Конвергенция генов и клеточных путей, нарушающих регуляцию при расстройствах аутистического спектра». Американский журнал генетики человека. 94 (5): 677–694. Дои:10.1016 / j.ajhg.2014.03.018. ЧВК 4067558. PMID 24768552.
- ^ Леви, Дэн; Ронемус, Майкл; Ямром, Борис; Ли, Юн-ха; Леотта, Энтони; Кендалл, Джуд; Маркс, Стивен; Лакшми, Б .; Пай, Дипа; Йе, Кенни; Буя, Андреас; Кригер, Абба; Юн, Сынтай; Троге, Дженнифер; Роджерс, Линда; Иосифов, Иван; Виглер, Майкл (Июнь 2011 г.). «Редкое De Novo и изменение числа передаваемых копий при расстройствах аутистического спектра». Нейрон. 70 (5): 886–897. Дои:10.1016 / j.neuron.2011.05.015. PMID 21658582. S2CID 11132936.
- ^ Сандерс, Стефан Дж .; Эрджан-Сеничек, А. Гюльхан; Хас, Ванесса; Луо, Руи; Мурта, Майкл Т .; Морено-Де-Лука, Даниэль; Chu, Su H .; Моро, Майкл П .; Gupta, Abha R .; Thomson, Susanne A .; Мейсон, Кристофер Э .; Билгувар, Кая; Селестино-Сопер, Патриция Б.С.; Чой, Мурим; Кроуфорд, Эмили Л .; Дэвис, Ли; Дэвис Райт, Николь Р .; Dhodapkar, Rahul M .; ДиКола, Майкл; ДиЛулло, Николас М .; Фернандес, Томас V .; Филдинг-Сингх, Викрам; Фишман, Дэниел О .; Фрам, Стефани; Гарагалоян, Рубен; Го, Джеральд С .; Каммела, Синдхуджа; Клей, Ламбертус; Лоу, Дженнифер К .; Lund, Sabata C .; МакГрю, Анна Д .; Мейер, Кайл А .; Моффат, Уильям Дж .; Мердок, Джон Д .; О'Роак, Брайан Дж .; Обер, Гордон Т .; Pottenger, Rebecca S .; Raubeson, Melanie J .; Песня, Youeun; Ван, Ци; Яспан, Брайан Л .; Ю, Тимоти В .; Юркевич, Илана Р .; Beaudet, Arthur L .; Кантор, Рита М .; Керленд, Мартин; Грайс, Дороти Э .; Гюнель, Мурат; Лифтон, Ричард П .; Mane, Shrikant M .; Мартин, Донна М .; Shaw, Chad A .; Шелдон, Майкл; Tischfield, Jay A .; Уолш, Кристофер А .; Morrow, Eric M .; Ледбеттер, Дэвид Х .; Фомбонн, Эрик; Лорд, Екатерина; Мартин, Криста Лезе; Брукс, Эндрю I .; Сатклифф, Джеймс С .; Кук, Эдвин Х .; Гешвинд, Даниэль; Рёдер, Кэтрин; Девлин, Берни; Государство, Мэтью В. (июнь 2011 г.). «Множественные рецидивирующие CNV De Novo, включая дублирование области синдрома Вильямса 7q11.23, прочно связаны с аутизмом». Нейрон. 70 (5): 863–885. Дои:10.1016 / j.neuron.2011.05.002. ЧВК 3939065. PMID 21658581.
- ^ Джонсон, Мэтью Э .; Виджиано, Луиджи; Бейли, Джеффри А .; Абдул-Рауф, Мунах; Гудвин, Грэм; Рокки, Мариано; Эйхлер, Эван Э. (2001). «Положительный отбор семейства генов во время появления человека и африканских обезьян». Природа. 413 (6855): 514–9. Bibcode:2001Натура.413..514J. Дои:10.1038/35097067. PMID 11586358. S2CID 4327069.
- ^ Редон, Ричард; Исикава, Шумпей; Fitch, Karen R .; Феук, Ларс; Перри, Джордж Х .; Эндрюс, Т. Дэниэл; Fiegler, Heike; Шаперо, Майкл Х .; и другие. (2006). «Глобальные вариации числа копий в геноме человека». Природа. 444 (7118): 444–54. Bibcode:2006Натура.444..444R. Дои:10.1038 / природа05329. ЧВК 2669898. PMID 17122850.
- ^ Gonzalez, E .; Kulkarni, H; Боливар, H; Mangano, A; Санчес, Р. Catano, G; Nibbs, RJ; Фридман, Б.И.; и другие. (2005). «Влияние дупликаций сегментов, содержащих ген CCL3L1, на восприимчивость к ВИЧ-1 / СПИДу». Наука. 307 (5714): 1434–40. Bibcode:2005Sci ... 307.1434G. Дои:10.1126 / science.1101160. PMID 15637236.
- ^ Bubb, K. L .; Bovee, D; Бакли, Д; Haugen, E; Кибукава, М; Паддок, М; Palmieri, A; Субраманиан, S; и другие. (2006). «Сканирование генома человека не обнаруживает новых локусов при древнем сбалансированном отборе». Генетика. 173 (4): 2165–77. Дои:10.1534 / genetics.106.055715. ЧВК 1569689. PMID 16751668.
- ^ "Human hg38 chr1: 11 102 837-11 267 747 UCSC Genome Browser v374".
- ^ «Обзор структурных изменений».
- ^ а б Таттини, Лоренцо; Д'Аурицио, Ромина; Маги, Альберто (2015). «Обнаружение структурных вариантов генома на основе данных секвенирования следующего поколения». Границы биоинженерии и биотехнологии. 3 (92): 92. Дои:10.3389 / fbioe.2015.00092. ЧВК 4479793. PMID 26161383.
- ^ а б c d е Feuk, L .; Carson, A.R .; Schere, S.W. (2006). «Структурные вариации в геноме человека». Природа Обзоры Генетика. 7 (2): 85–97. Дои:10.1038 / nrg1767. PMID 16418744. S2CID 17255998.
- ^ Алкан, банка; Коу, Брэдли П.; Эйхлер, Эван Э. (2011). «Открытие структурных вариаций генома и генотипирование». Природа Обзоры Генетика. 12 (5): 363–376. Дои:10.1038 / nrg2958. ЧВК 4108431. PMID 21358748.
- ^ Кузняр, Арнольд; Маассен, Джейсон; Верховен, Стефан; Сантуари, Лука; Шнейдер, Карл; Kloosterman, Wigard P .; де Риддер, Джерун (2020). "sv-callers: портативный параллельный рабочий процесс для обнаружения структурных вариантов в данных последовательности всего генома". PeerJ. 8 (5): 2167–8359. Дои:10.7717 / peerj.8214. ЧВК 6951283. PMID 31934500.
