Малая ядрышковая РНК U3 - Small nucleolar RNA U3
| U3 | |
|---|---|
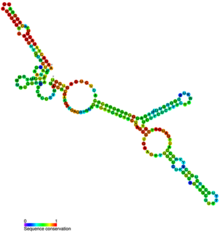 Вторичная структура РНК U3 метазоа и сохранение последовательности | |
| Идентификаторы | |
| Символ | U3 |
| Альт. Символы | РНУ3П2, |
| Рфам | RF00012 |
| NCBI Gene | 26844 |
| HGNC | 10176 |
| OMIM | 180710 |
| Прочие данные | |
| РНК тип | snoRNA |
| Домен (ы) | Эукариоты; |
| PDB структуры | PDBe |
U3 мяРНК это некодирующая РНК находится преимущественно в ядрышко.U3 имеет Мотивы коробки C / D которые технически делают его членом коробочного класса C / D snoRNAs; однако, в отличие от других мяноРНК C / D-бокса, не было показано, что он направляет 2'-О-метилирование других РНК, скорее считается, что U3 управляет сайт-специфическим расщеплением рибосомная РНК (рРНК) во время обработки пре-рРНК.[1]
Элемент C / D бокса представляет собой подмножество шести элементов короткой последовательности, обнаруженных во всех snoRNA U3, а именно боксов A, A ', B, C, C' и D.[2] МнорНК U3 вторичная структура характеризуется небольшой 5' домен (с полями A и A ') и больший 3' домен (с блоками B, C, C 'и D), причем два домена связаны однонитевым шарниром. Боксы B и C образуют мотив B / C, который, по-видимому, является эксклюзивным для snoRNA U3, а боксы C 'и D образуют мотив C' / D. Последний функционально подобен мотивам C / D, обнаруженным в других мяРНК. 5'-домен и шарнирная область действуют как пре-рРНК-связывающий домен. 3'-домен имеет консервативные сайты связывания с белками. Мотивы как бокса B / C, так и бокса C '/ D достаточны для удержания в ядре мяРНК U3. Мотив C '/ D бокс также необходим для локализации ядрышка, стабильности и гиперметилирования U3 snoRNA.[3] Мотивы бокса B / C и C '/ D вовлечены в специфические белок взаимодействия и необходимы для рРНК процессинговые функции мяРНК U3.
Видовые модели вторичной структуры
С. cerevisiae вторичная структура, определенная химическим картированием РНК U3A в очищенном snoRNP, доступна.[4] А человек Также была предложена структурная модель.[5] Подобно дрожжам и человеку, простейший протист Entamoeba histolytica: примитивный эукариот принял такую же консервативную вторичную структуру мяРНК U3.[6] Четыре структуры консенсуса, характерные для метазоа, грибы, растения и базальные эукариоты.[7]
Смотрите также
Рекомендации
- ^ Cléry, A .; Senty-Ségault, V .; Leclerc, F .; Raué, A .; Бранлант, К. (февраль 2007 г.). «Анализ последовательности и структурных особенностей, которые идентифицируют мотив B / C малой ядрышковой РНК U3 в качестве сайта узнавания для пары белков Snu13p-Rrp9p». Молекулярная и клеточная биология. 27 (4): 1191–1206. Дои:10.1128 / MCB.01287-06. ISSN 0270-7306. ЧВК 1800722. PMID 17145781.
- ^ Цвиб, С. (1997). «База данных уРНК». Нуклеиновые кислоты Res. 25 (1): 102–103. Дои:10.1093 / nar / 25.1.102. ЧВК 146409. PMID 9016512.
- ^ Спекманн, Вт; Нараянан А; Terns R; Крачка М.П. (1999). «Ядерные удерживающие элементы малой ядрышковой РНК U3». Mol Cell Biol. 19 (12): 8412–8421. Дои:10.1128 / MCB.19.12.8412. ЧВК 84939. PMID 10567566.
- ^ Меро А., Фурнье Р., Грегуар А. и др. (Октябрь 1997 г.). «Анализ структуры-функции in vivo и in vitro snoRNP U3A Saccharomyces cerevisiae: контакты белок-РНК и взаимодействие пар оснований с прерибосомной РНК». J. Mol. Биол. 273 (3): 552–71. Дои:10.1006 / jmbi.1997.1320. PMID 9356246.
- ^ Граннеман С., Фогельзангс Дж., Люрманн Р., ван Венроой В.Дж., Пруейн Г.Дж., Уоткинс Нью-Джерси (октябрь 2004 г.). «Роль спаривания оснований пре-рРНК и образование комплекса 80S в субнуклеолярной локализации snoRNP U3». Мол. Клетка. Биол. 24 (19): 8600–10. Дои:10.1128 / MCB.24.19.8600-8610.2004. ЧВК 516741. PMID 15367679.
- ^ Шривастава А., Ахамад Дж., Рэй А.К., Каур Д., Бхаттачарья А., Бхаттачарья С. (2014). Анализ U3 snoRNA и компонентов процессома малой субъединицы у паразитического протиста Entamoeba histolytica. Мол Биохим Паразитол. 193 (2): 82-92. DOI: 10.1016 / j.molbiopara.2014.03.001. Epub 2014 12 марта
- ^ Марз М, Штадлер П.Ф. (2009). «Сравнительный анализ snoRNA U3 эукариот». РНК Биол. 6 (5): 503–7. CiteSeerX 10.1.1.380.4189. Дои:10.4161 / rna.6.5.9607. PMID 19875933.
внешняя ссылка
- Страница для Малой ядрышковой РНК U3 в Рфам
- uRNADB: страница U3 (архив)
- Запись UMASS snoRNAdb для U3
- Запись в SGD для U3a
- Запись человеческой snoRNAbase для U3
| Этот молекулярная биология статья - это заглушка. Вы можете помочь Википедии расширяя это. |
